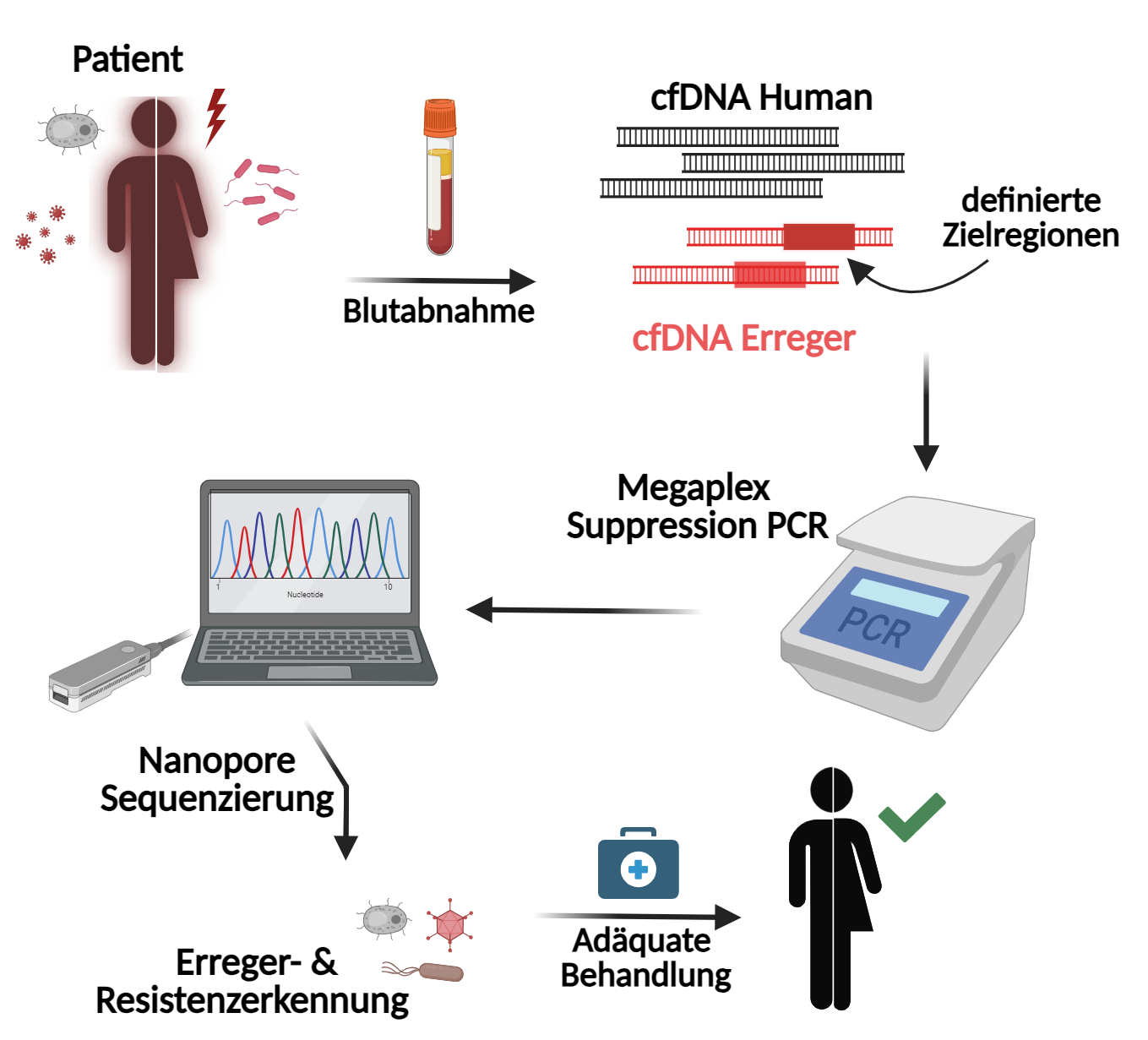
Mikrobielle Resistenzen gegen Antibiotika sind ein weltweites Problem und ihre Diagnose von großer Bedeutung, um die effektivste Behandlung von Infektionen zu bestimmen und die Ausbreitung von resistenten Organismen einzudämmen.
Aktuell umfassen Labortests aufwändige mikro- bzw. zellbiologische Analysen, die zeit- und kostenintensiv sind. Aufgrund der Gefahr von Kontaminationen und ihrer geringen Genauigkeit werden dringend neue Methoden zur Erkennung benötigt.
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB