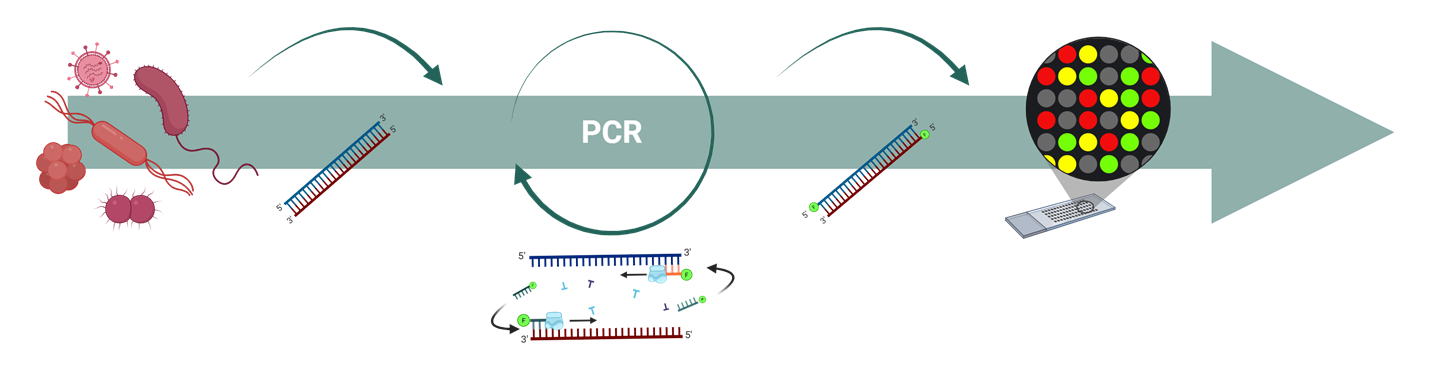
Mit langjähriger Erfahrung entwickeln wir auf Nukleinsäuren beruhende Microarrays zur molekularen Diagnostik einer Vielzahl von Erregern. Unsere Aktivitäten umfassen den gesamten Workflow der Array-Entwicklung vom Sondendesign, über deren Immobilisierung im Kontaktprintverfahren bis hin zur Hybridisierung von Target-DNAs zur Fluoreszenz-basierten Identifikation der Erreger.
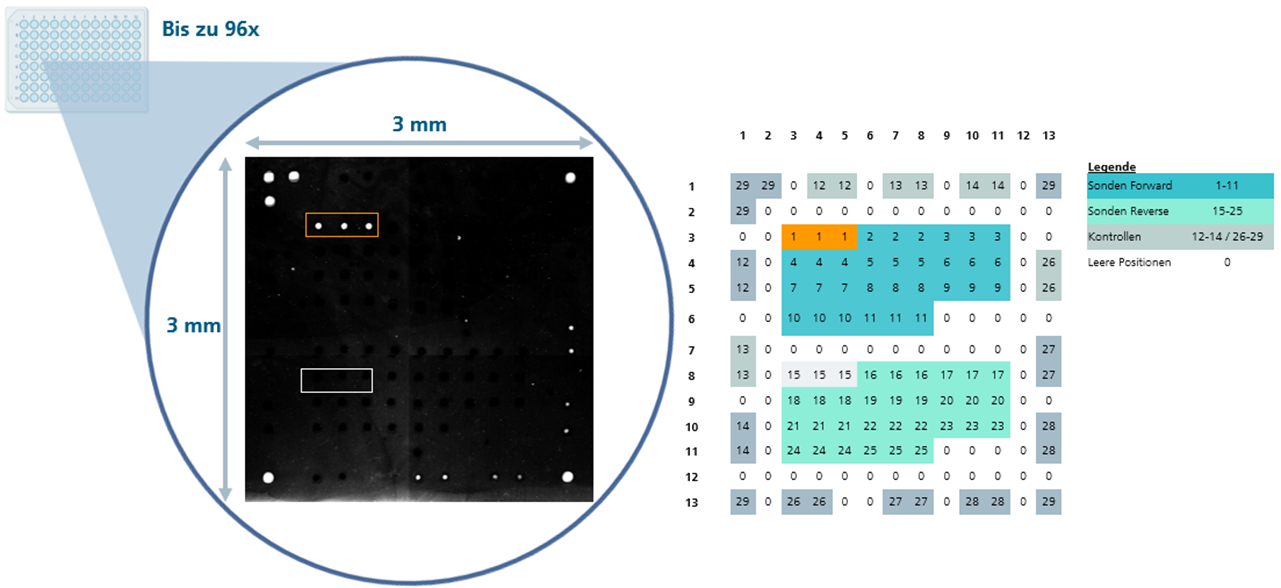
Das grundlegende Prinzip eines Microarrays oder Biochips ist die geordnete, punktförmige Immobilisierung von bis zu tausenden DNA-Sonden mit definierter Sequenz auf einem festen Träger.
Microarrays ermöglichen es so, eine Vielzahl von Genen bzw. Proteinen in einer einzigen biologischen Probe gleichzeitig zu untersuchen. Die einzelnen DNA/Protein-Spezies dienen dabei als Sensoren, die entsprechende Biomarker erkennen, um Aussagen über die Sequenz, Transkription, Expression oder auch Funktion dieser Gene und Proteine zu erhalten.
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB